Seebohms Tapuit
Oenanthe seebohmi · Seebohm's Wheatear
 Seebohms Tapuit ·
Oenanthe seebohmi
·
12-12-2018 · George Sangster
Seebohms Tapuit ·
Oenanthe seebohmi
·
12-12-2018 · George Sangster
| Datum | 12 december 2018 |
|---|---|
| Locatie | Marokko |
| Fotograaf |
|
| Bekeken | 5742 × |
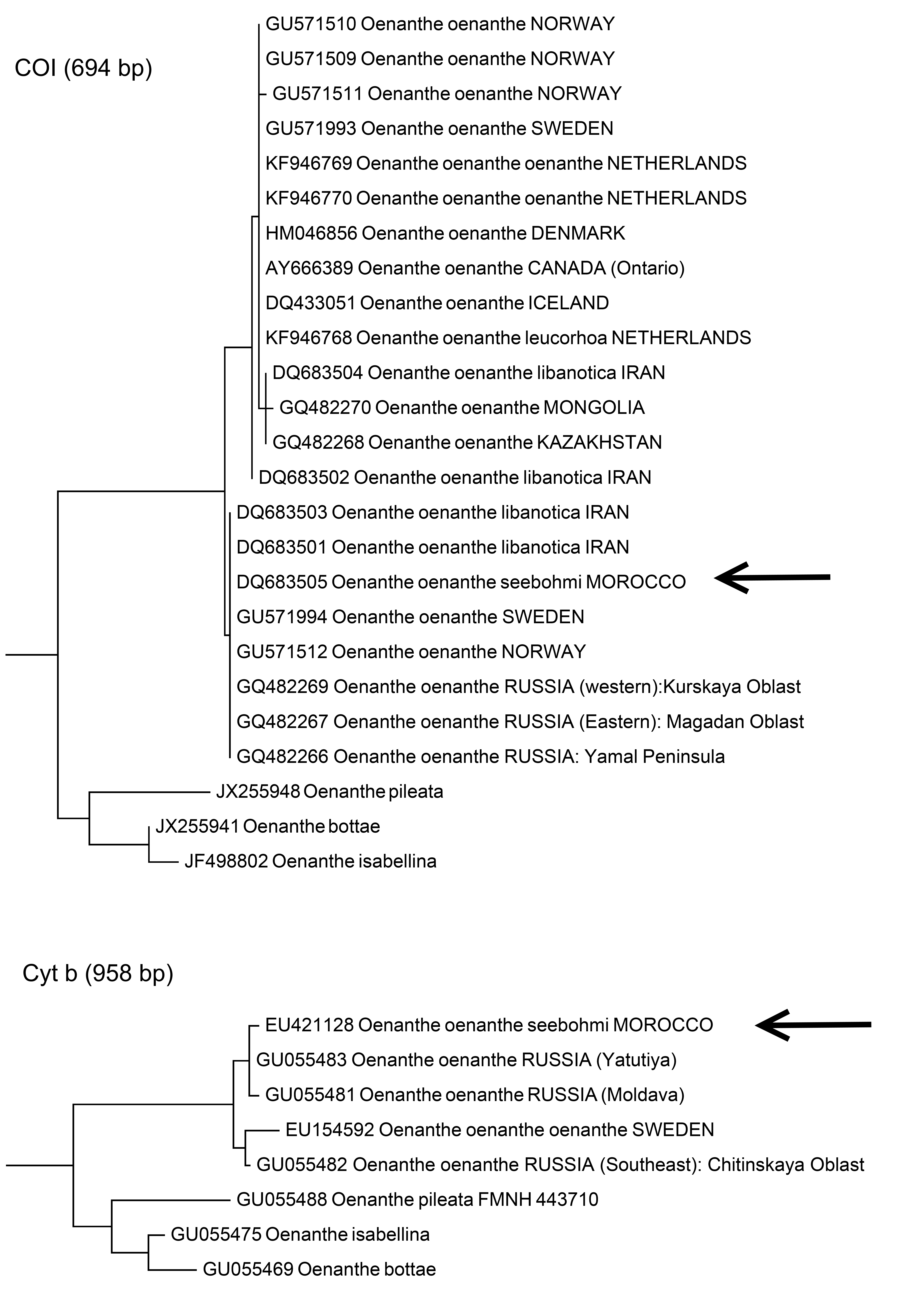
Twee mtDNA stambomen, gemaakt met behulp van GenBank sequenties. Het gaat om de genen cytochrome c oxidase subunit I (CO1, a.k.a. het 'DNA barcoding' gen) en cytochroom b. (Outgroepen zijn niet weergegeven.) In beide gevallen valt de sample van een Marokkaanse Seebohms Tapuit binnen die van gewone Tapuiten, waarbij de verschillen tussen de twee taxa minimaal zijn. De sequenties van Seebohms Tapuit zijn door twee verschillende onderzoeksgroepen gepubliceerd (Aliabadian et al. 2007, Illera et al. 2008). De figuur impliceert dat mtDNA identificatie van Seebohms Tapuit met behulp van het huidige vergelijkingsmateriaal niet mogelijk is. |
Discussie
Jelle Scharringa
·
13 december 2018 08:59
Veren verzamelen dus!
Rob Poot
·
10 januari 2019 20:37
George, als er bij het sneller muterende vrouwelijk genetisch materiaal nog weinig te zien is, wat is te verwachten bij mannelijk genetisch materiaal?
Of is daar nog niets van te zeggen?
Peter de Knijff
·
10 januari 2019 21:15, gewijzigd 10 januari 2019 21:16
Rob,
Dit is op geen enkele manier betrouwbaar te voorspellen omdat het van te veel nogal fundamenteel genetische, evolutionaire en populatie demografische aspecten afhangt.
Geen scenario is zo gek of het is al waargenomen.
We moeten gewoon geduld hebben tot veel meer genetische gegevens van deze tapuitengroep beschikbaar komen. Dat komt heus wel, deze taxa zijn razend interessante metabolisme studie modellen en er wordt op dit moment erg veel genetisch onderzoek naar gedaan (in Duitsland).
gr.
Peter
Rob Poot
·
12 januari 2019 16:42
Dank je Peter, gewoon rustig afwachten dus.
Justin Jansen
·
23 juli 2020 07:38
Wang, E., Zhang, D., Braun, M.S. et al. Can Mitogenomes of the Northern Wheatear (Oenanthe oenanthe) Reconstruct Its Phylogeography and Reveal the Origin of Migrant Birds?. Sci Rep 10, 9290 (2020). https://doi.org/10.1038/s41598-020-66287-0
Abstract:
The Northern Wheatear (Oenanthe oenanthe, including the nominate and the two subspecies O. o. leucorhoa and O. o. libanotica) and the Seebohm’s Wheatear (Oenanthe seebohmi) are today regarded as two distinct species. Before, all four taxa were regarded as four subspecies of the Northern Wheatear. Their classification has exclusively been based on ecological and morphological traits, while their molecular characterization is still missing. With this study, we used next-generation sequencing to assemble 117 complete mitochondrial genomes covering O. o. oenanthe, O. o. leucorhoa and O. seebohmi. We compared the resolution power of each individual mitochondrial marker and concatenated marker sets to reconstruct the phylogeny and estimate speciation times of three taxa. Moreover, we tried to identify the origin of migratory wheatears caught on Helgoland (Germany) and on Crete (Greece). Mitogenome analysis revealed two different ancient lineages that separated around 400,000 years ago. Both lineages consisted of a mix of subspecies and species. The phylogenetic trees, as well as haplotype networks are incongruent with the present morphology-based classification. Mitogenome could not distinguish these presumed species. The genetic panmixia among present populations and taxa might be the consequence of mitochondrial introgression between ancient wheatear populations.
Gebruikers van het forum gaan akkoord met de forumregels.
